什么样的进阶武器,会有那么大威力?
CUT&Tag——ChIP-Seq进阶武器
一、染色质免疫沉淀后测序(ChIP-Seq)
ChIP-Seq是一种针对DNA结合蛋白、组蛋白修饰或者核小体的全基因组测序分析技术。其基本原理是利用甲醛处理细胞,使目标蛋白与DNA交联,通过超声破碎将交联后的染色质打碎成小片段,一般在200-600bp。再利用抗原-抗体特异性识别反应,将目的蛋白连同交联的DNA片段一起沉淀下来。洗脱蛋白质沉淀,对纯化后的DNA进行PCR扩增,高通量测序,最后与已有的基因组序列进行比对,以确定与目的蛋白交联的DNA的序列。
ChIP-Seq可以完整的反映与靶蛋白结合的DNA序列,是蛋白质与DNA互作的经典方法。但是常规的ChIP-Seq需要大量的细胞样本,而且对培养环境、处理条件要求较高,且实验重复性较差、信噪比低。很多时候会发现,培养了很长周期的细胞,经过ChIP-Seq测序后得到的却是大量的垃圾数据,这些限制了ChIP-Seq的应用,亟需进化升级。
二、CUT&Tag(Cleavage Under Targets and Tagment)技术
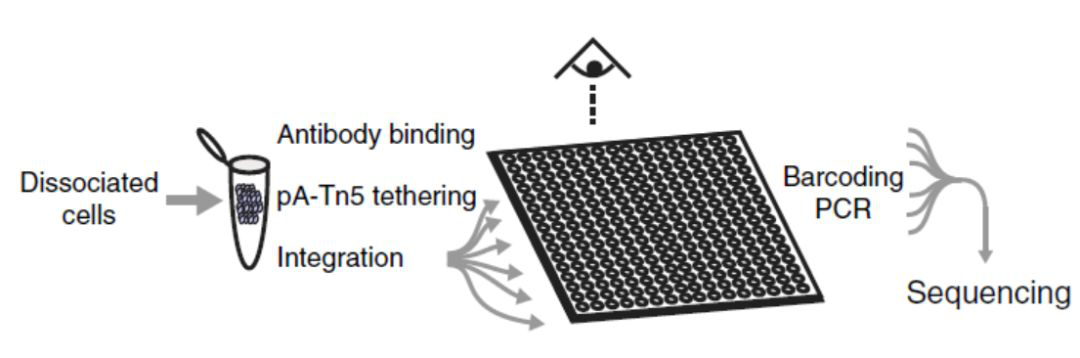
2019年4月底,弗雷德哈钦森癌症研究中心的Henikoff博士,在Nature Communication杂质上公开了CUT&Tag技术的详细结果和实验方案。能在全基因层面上检测与组蛋白、转录因子等相互作用的DNA片段。这种方法相较于ChIP-Seq、pA-MNase等方法,更加简便,信噪比更高、重复性更好、需要的细胞数量更少,切有希望将蛋白质-DNA结合研究做到单细胞水平。
CUT&Tag方法是Active motif专利技术TAM-ChIP技术的一个变形。CUT&Tag是基于ChIP原理的一项技术,与ChIP的免疫沉淀步骤相比,同样是利用抗原抗体结合,但是CUT&Tag是抗体孵育后直接剪切染色质和制备文库。
在CUT&Tag实验中,细胞首先要经过透化处理,并将其与刀豆蛋白A一起孵育,以便于后续清洗步骤。细胞随后先后与特异性抗体(一抗、二抗)孵育;然后将细胞与转座体孵育,转座体由带有NGS接头(adaptor)的Tn5转座酶和蛋白A(protein A)融合而成。清洗掉未结合的转座体。Tn5转座酶是Mg
2+依赖的酶,因此需要加入Mg
2+以激活反应,从而使染色质被切割到接近蛋白结合位点的位置,同时链接上NGS测序接头DNA序列。这使得染色质裂解和文库制备只需一步完成。
三、
CUT&Tag具体实验方案
1. 细胞预处理(0.5-1h)
1) 室温下收集新鲜细胞置于EP管中并计数;低速离心,去溶液;
2) 管中加入细胞穿孔缓冲液重悬细胞;低速离心,去溶液;
3)管中加入细胞穿孔缓冲液再重悬细胞,轻柔震荡并滴加Binding 缓冲液重悬的ConA磁珠,轻柔颠倒混匀5-10min;
2. 一抗结合目的蛋白(2h)
1) 快速离心EP管,将管盖液体离心下来,将EP管静置于磁力架上沉淀细胞,去溶液。
2) EP管中加入预冷的抗体缓冲液将细胞重悬,轻柔震荡后置于冰上;
3) 每管中加入0.5-1μl一抗(抗体使用生产商推荐浓度),轻柔震荡;
4) 室温下将EP管置于摇床上孵育2h;
3. 二抗结合一抗(1h)
1) 快速离心EP管,将管盖液体离心下来,将EP管静置于磁力架沉淀细胞,去溶液。
2) 将二抗1:100稀释于穿孔缓冲液,加入管中,轻柔震荡;
3) 室温下将EP管置于摇床上孵育30-60min;
4) 快速离心细胞,将管盖液体离心下来,EP管静置于磁力架沉淀细胞,去溶液。
5) 管中加入0.8-1ml 穿孔缓冲液,上下颠倒10次;
6) 重复步骤4)、5) 两次。
4. ChiTag转座体结合抗体(1.5h)
1) 快速离心EP管,将管盖液体离心下来,将EP管静置于磁力架沉淀细胞,去溶液。
2) 将ChiTag转座体1:250稀释于穿孔缓冲液2中,并滴加于细胞上,轻柔震荡;
3) 室温下将EP管置于摇床上孵育1h;
4) 快速离心细胞,将管盖液体离心下来,将EP管静置于磁力架沉淀细胞,去溶液。
5) 管中加入0.8-1ml穿孔缓冲液2,上下颠倒10次;
6) 重复步骤4)、5) 两次。
5. 激活ChiTag转座体,片段化目的DNA(1h)
1) 快速离心EP管,将管盖液体离心下来,将EP管静置于磁力架沉淀细胞,去溶液。
2) 管中加入300μl ChiTag激活缓冲液,轻柔震荡;
3) 37ºC孵育1h。
6. DNA提取(1h)
7. PCR(1h),PCR之前需加入72ºC,5min步骤
8. DNA纯化(30min)
9. 上机测序
具体时间、转速需要根据不同的样本、不同的研究目的进行优化。
四、CUT&Tag的局限性
1. CUT&Tag的自然状态并不适合所有研究
未经固定的细胞不适用CUT&Tag。在CUT&Tag最初的文章中,此技术只用于组蛋白修饰、NPAT、CTCF。许多转录因子并不大量表达,且与DNA结合是微弱的、瞬时的,甚至有时候是间接的与染色质结合。对于这些情况,染色质交联和超声处理是检测蛋白质-DNA相互作用的必要步骤。
大多数ChIP级别抗体,经验证可用于交联条件下,然而无法在自然情况下具有好的作用,因为交联条件下的蛋白质表位可能不同于自然状态下的蛋白质表位。因此从X-ChIP转到CUT&Tag需要进行抗体验证,以确定抗体在不固定的条件下的特异性和敏感性。
2. CUT&Tag可能引入偏差
用于CUT&Tag的Tn5转座酶对开放染色质区域具有高度亲和性。因此,CUT&Tag可能优先适用于分析组蛋白修饰或与基因组活跃转录区域相关转录因子,而不是非常适合于沉默或含有异染色质的基因组区域。消化时间和pA-Tn5使用量需要每个目标仔细优化,以避免任何不特异的转座反应。
3. CUT&Tag技术还太新
虽然CUT&Tag已经出现了有一段时间了,且很快就流行了起来。但是在2019年4月才首次发表详细的实验方案。因此,在同行评议中并没有很多包含CUT&Tag结果的数据出现。另外,CUT&Tag实验流程与ChIP-Seq相差很大,两者的数据对比也比较困难,
 吉ICP备2021001101号-1
吉ICP备2021001101号-1